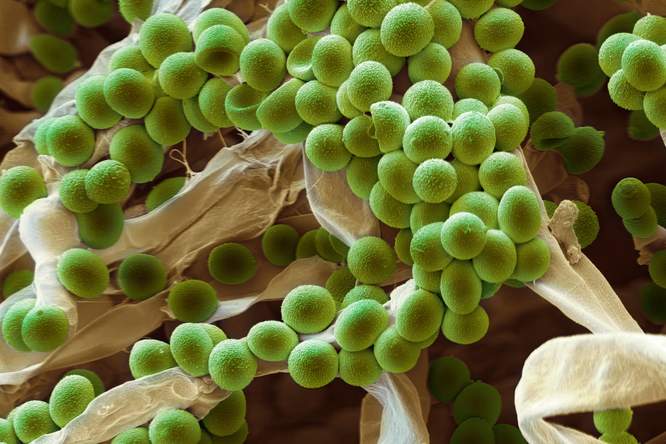
Zoeken naar nieuwe vormen van antibiotica is als zoeken naar een speld in een hooiberg. Een computeralgoritme moet dit proces versnellen en stroomlijnen.
'Antibiotica kunnen overal voorkomen: in de bodem, lucht, rivieren en meren en zelfs in zee – overal dus waar bacteriën voorkomen'
Peptiden zijn opgebouwd uit een klein aantal aminozuren. Op hun beurt zijn aminozuren de bouwstenen van eiwitten. De microbiologische componenten bestaan in ontelbaar veel varianten. Sommige varianten die in de natuur vrij voorkomen hebben bovendien een antibiotische werking – ze doden specifieke bacteriën. Nu de voorraad beschikbare effectieve antibiotica slinkt, zijn medische onderzoekers volop bezig met de zoektocht naar nieuwe stoffen.
Naar potentieel nieuwe antibiotica speuren is als zoeken naar een speld in een hooiberg. Ze kunnen overal voorkomen: in de bodem, lucht, rivieren en meren en zelfs in zee – overal dus waar bacteriën voorkomen, die ze immers produceren. Een analyse van een staal levert dus een gigantische hoeveelheid data – big data – op. Zelfs met krachtige computers is het niet simpel om beloftevolle peptiden te vinden.
Een team van Amerikaanse en Russische wetenschappers heeft een algoritme ontwikkeld waarmee ze, veel sneller dan vroeger, varianten van reeds bestaande antibiotica kunnen opsporen. Tijdens een demonstratie bleek de computerinstructie slechts een paar uur nodig te hebben om de volledige, huidig bekende peptidedatabase tevoorschijn te toveren.
Als een bacterie resistent is tegen een welbepaald antibioticum, is ze dat vaak ook tegen de varianten erop. De onderzoekers willen hun algoritme daarom gaan stroomlijnen en vervolgens loslaten op willekeurige bodemstalen waarin nog geen gekende antibiotica zijn gevonden.